Estudantes e profissionais da área química precisam realizar diversos cálculos no dia a dia. Tais cálculos muitas vezes são complexos devido à grande quantidade de dados e de aproximações. Ferramentas computacionais facilitam a resolução dessas atividades.
Nesse artigo, vamos explorar 3 bibliotecas químicas disponíveis para a linguagem Python, mostrando como elas são úteis na nossa rotina.
Tópicos
Molmass
Começamos nosso Top 3 com o Molmass, um pacote Python que permite calcular a massa molar de uma forma bem simples. Ao invés de calcular tediosamente as massas molares de compostos somando as dos seus elementos constituintes, basta passar sua fórmula, como veremos.
A instalação pode ser feita via pip install molmass. E, para importar:
from molmass import FormulaVamos começar com um exemplo simples: cerveja! Mais especificamente, o etanol presente nas cervejas.
Podemos calcular a massa molar do etanol facilmente com o Molmass. Para isso, passamos uma string com a fórmula do etanol para a classe Formula e solicitamos o atributo mass, a unidade utilizada é g/mol, como informado no comentário:
formula = Formula('C2H5OH')
formula.mass # g/mol46.068531
Podemos observar que o pacote utiliza as massas molares sem arredondamentos, o que é excelente, pois o uso de aproximações leva à propagação de erros.
Ah, e se quiser continuar o papo de cerveja, veja como ela já deu origem à uma biblioteca Python nesse artigo. Mas só depois de terminar este artigo aqui 🙂
O Molmass aceita diferentes entradas para um mesmo composto. Vamos passar outra string com a abreviação clássica do radical etil na orgânica, Et, para obter a massa molar do etanol:
formula = Formula('EtOH')
formula.mass46.068531
Observe que as fórmulas diferenciam maiúsculas de minúsculas.
Para obter informações da composição elementar do composto, podemos utilizar o método composition:
formula.composition()(('C', 2, 24.02148, 0.5214292593788155),
('H', 6, 6.047646, 0.13127499116479316),
('O', 1, 15.999405, 0.34729574945639136))
Vemos que o método composition fornece colunas com os atributos element, number, relative_mass e fraction, respectivamente.
Mendeleev
Outro pacote Python muito útil no nosso dia a dia é o Mendeleev. Ele permite acessar várias propriedades de elementos, íons e isótopos.
O pacote pode ser instalado via pip install mendeleev.
Tabela periódica
Uma das caracteristicas mais legais do Mendeleev é a possibilidade de importar uma tabela periódica com apenas uma linha de código. Para isso precisamos ter a dependência vis, que pode ser instalada via pip install mendeleev[vis].
Uma maneira de visualizar a tabela periódica é usando a função periodic_table do módulo mendeleev.vis:
from mendeleev.vis import periodic_tablePara observar a visualização padrão da tabela periódica vamos chamar a função importada:
periodic_table()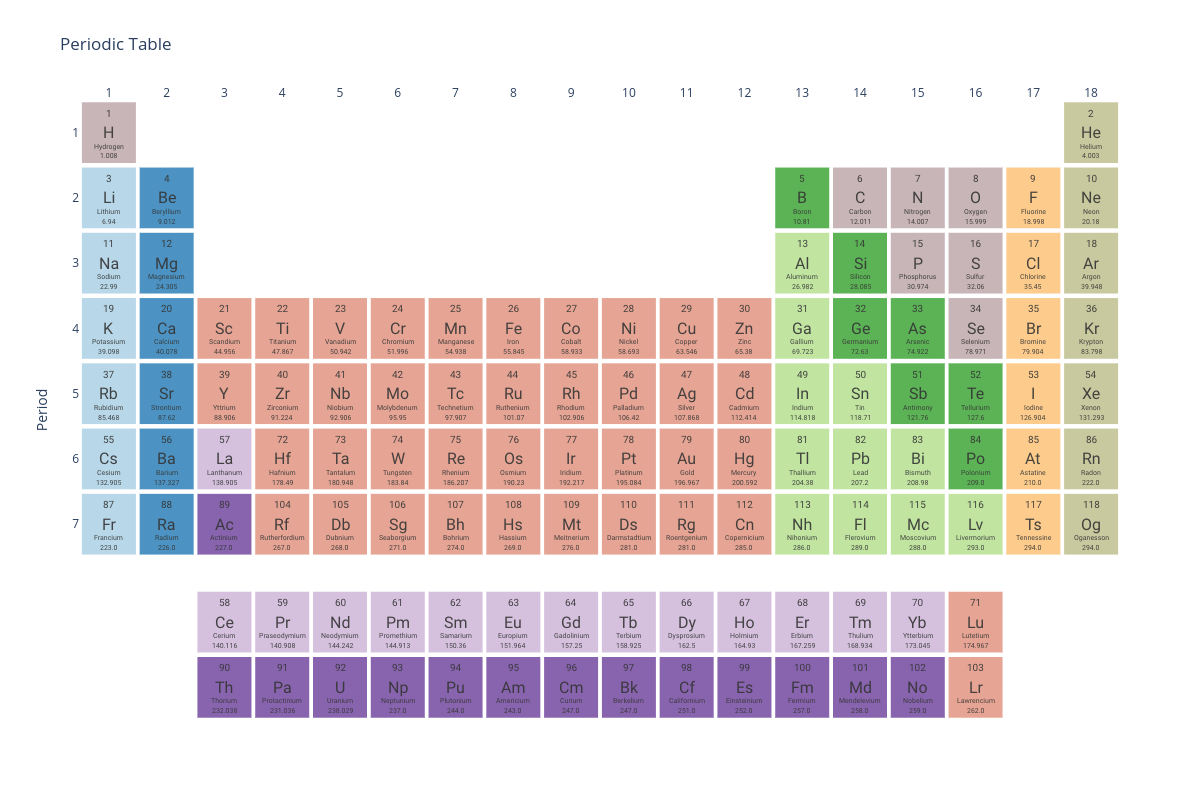
Propriedades de elementos
Agora de um ponto de vista mais técnico. Para acessar os dados de um elemento basta importar por meio de seus símbolos. Por exemplo, considere o cromo:
from mendeleev import CrPara extrair alguma informação podemos solicitar um atributo, por exemplo, o boiling_point onde vamos obter a temperatura de ebulição em Kelvin, como informado no comentário:
Cr.boiling_point # K2945.0
Uma forma alternativa para acessar os dados é por meio do element, método que retorna uma única instância ou uma tupla dessas instâncias, dependendo dos argumentos:
from mendeleev import elementO método element aceita o nome do elemento em inglês, o símbolo atômico ou o número atômico. Vejamos um exemplo em que passamos o número atômico do bromo para o método element e solicitamos o atributo atomic_radius onde vamos obter o raio atômico em picometros, como informado no comentário:
bromo = element(35)
bromo.atomic_radius # pm115.0
O método element também aceita lista ou tupla. Observe esse exemplo em que passamos uma lista para o método element e solicitamos o atributo econf onde vamos obter a configuração eletronica dos elementos:
hidrogenio, zinco, bromo = element(['Hydrogen', 'Zn', 35])
hidrogenio.econf, zinco.econf, bromo.econf('1s', '[Ar] 3d10 4s2', '[Ar] 3d10 4s2 4p5')
Com o Mendeleev também conseguimos obter a configuração eletrônica do íon a partir do elemento. Tomemos como exemplo o íon Fe3+. Primeiramente, passamos o nome do elemento em inglês para o método element e solicitamos o atributo ec onde vamos obter a configuração eletrônica do átomo de ferro:
ferro = element('Iron')
ferro.ec<ElectronicConfiguration(conf="1s2 2s2 2p6 3s2 3p6 3d6 4s2")>
Para obter a configuração eletrônica do íon Fe3+ vamos utilizar o método ionize a fim de remover os 2 elétrons da última camada eletrônica e não do subnível mais energético e 1 elétron, agora sim, do subnível mais energético, retornando a seguinte configuração eletrônica:
ferro.ec.ionize(3)<ElectronicConfiguration(conf="1s2 2s2 2p6 3s2 3p6 3d5")>
Existem alguns atributos como o isotopes que retorna uma lista de objetos com as propriedades de cada isótopo atomic_number, mass_number, mass e abundance, respectivamente:
enxofre = element('S')
for iso in enxofre.isotopes:
print(iso)16 32 31.97207 0.944 16 33 32.97146 0.008 16 34 33.96787 0.048 16 36 35.96708 0.000
O ionic_radii retorna uma lista de objetos com os atributos charge, coordination, crystal_radius em pm, e ionic_radius em pm:
ouro = element('Au')
for ir in ouro.ionic_radii:
print(ir)charge= 1, coordination=VI , crystal_radius=151.000, ionic_radius=137.000 charge= 3, coordination=IVSQ , crystal_radius=82.000, ionic_radius=68.000 charge= 3, coordination=VI , crystal_radius=99.000, ionic_radius=85.000 charge= 5, coordination=VI , crystal_radius=71.000, ionic_radius=57.000
O ionenergies retorna um dicionário com energias de ionização:
oxigenio = element('O')
oxigenio.ionenergies{1: 13.618054,
2: 35.12111,
3: 54.93554,
4: 77.4135,
5: 113.8989,
6: 138.1189,
7: 739.32679,
8: 871.40985}
Outro atributo é o oxistates que retorna uma lista dos estados de oxidação mais comuns para um determinado elemento:
manganes = element('Mn')
manganes.oxistates[7, 6, 4, 3, 2]
O método element possui vários outros atributos e/ou métodos que nos permitem acessar diversas propriedades dos elementos. Vamos observar todos os disponíveis no objeto manganes que criamos:
manganes = element('Mn')
[x for x in dir(manganes) if not x.startswith('_')]['abundance_crust', 'abundance_sea', 'annotation', 'atomic_number', 'atomic_radius', 'atomic_radius_rahm', 'atomic_volume', 'atomic_weight', 'atomic_weight_uncertainty', 'block', 'boiling_point', 'c6', 'c6_gb', 'cas', 'covalent_radius', 'covalent_radius_bragg', 'covalent_radius_cordero', 'covalent_radius_pyykko', 'covalent_radius_pyykko_double', 'covalent_radius_pyykko_triple', 'cpk_color', 'density', 'description', 'dipole_polarizability', 'dipole_polarizability_unc', 'discoverers', 'discovery_location', 'discovery_year', 'ec', 'econf', 'electron_affinity', 'electronegativity', 'electronegativity_allen', 'electronegativity_allred_rochow', 'electronegativity_cottrell_sutton', 'electronegativity_ghosh', 'electronegativity_gordy', 'electronegativity_li_xue', 'electronegativity_martynov_batsanov', 'electronegativity_mulliken', 'electronegativity_nagle', 'electronegativity_pauling', 'electronegativity_sanderson', 'electronegativity_scales', 'electrons', 'electrophilicity', 'en_allen', 'en_ghosh', 'en_pauling', 'evaporation_heat', 'fusion_heat', 'gas_basicity', 'geochemical_class', 'glawe_number', 'goldschmidt_class', 'group', 'group_id', 'hardness', 'heat_of_formation', 'init_on_load', 'ionenergies', 'ionic_radii', 'is_monoisotopic', 'is_radioactive', 'isotopes', 'jmol_color', 'lattice_constant', 'lattice_structure', 'mass', 'mass_number', 'mass_str', 'melting_point', 'mendeleev_number', 'metadata', 'metallic_radius', 'metallic_radius_c12', 'molcas_gv_color', 'name', 'name_origin', 'neutrons', 'nvalence', 'oxides', 'oxistates', 'period', 'pettifor_number', 'proton_affinity', 'protons', 'registry', 'sconst', 'screening_constants', 'series', 'softness', 'sources', 'specific_heat', 'symbol', 'thermal_conductivity', 'uses', 'vdw_radius', 'vdw_radius_alvarez', 'vdw_radius_batsanov', 'vdw_radius_bondi', 'vdw_radius_dreiding', 'vdw_radius_mm3', 'vdw_radius_rt', 'vdw_radius_truhlar', 'vdw_radius_uff', 'zeff']
Propriedades de íons
O mendeleev possui a classe Ion para trabalhar com íons ao invés de elementos. Os íons podem ser criados a partir de elementos e informações de carga:
from mendeleev.ion import Ionferro_3 = Ion("Fe", 3)Vamos acessar algumas propriedades dos íons:
ferro_3.electrons23
ferro_3.name'Iron 3+ ion'
ferro_3.unicode_ion_symbol()'Fe³⁺'
ChemPy
ChemPy é um pacote Python que permite resolver problemas de química analítica, físico-química e inorgânica.
O pacote pode ser instalado via pip install chempy.
Balanceamento de equações
Com o ChemPy podemos balancear os coeficientes estequiométricos de uma reação. Para importar, simplesmente:
from chempy import balance_stoichiometryVamos imaginar um exemplo em que precisamos balancear a reação de combustão do acetileno, gás combustível empregado em maçaricos.

Para isso, passamos os reagentes e os produtos para a função balance_stoichiometry:
balance_stoichiometry({'C2H2', 'O2'}, {'CO', 'H2O'})(OrderedDict([('C2H2', 2), ('O2', 3)]), OrderedDict([('CO', 4), ('H2O', 2)]))
Obtivemos dois dicionários, um para os reagentes e outro para os produtos, com os coeficientes estequiométricos que balanceiam a equação.
Vamos agora considerar outro exemplo para mostrar que podemos obter a equação balanceada diretamente. Balancearemos a seguinte reação, que é utilizada em boosters de foguetes espaciais para auxiliar no lançamento dos mesmos:
NH_4ClO_4 + Al \rightarrow Al_2O_3 + HCl + H_2O + N_2reac, prod = balance_stoichiometry({'NH4ClO4', 'Al'}, {'Al2O3', 'HCl', 'H2O', 'N2'})reacOrderedDict([('Al', 10), ('NH4ClO4', 6)])
prodOrderedDict([('Al2O3', 5), ('H2O', 9), ('HCl', 6), ('N2', 3)])
Dessa maneira retorna um dicionário ordenado, solicitando a função dict criamos um dicionário padrão:
dict(reac){'Al': 10, 'NH4ClO4': 6}
dict(prod){'Al2O3': 5, 'H2O': 9, 'HCl': 6, 'N2': 3}
Podemos construir a equação balanceada usando a classe Reaction:
from chempy import Reactionreaction = Reaction(reac, prod)
reactionSistemas redox
Dentre os experimentos mais bonitos e interessantes de química temos as reações que mudam de cor. Esses experimentos de mudança de cor podem ocorrer, por exemplo, como resultado de reações de redução e oxidação.

No exemplo do GIF, a mudança de cor ocorre devido íons roxos de permanganato, MnO4–, receberem elétrons para formar íons incolores de manganês(II), Mn+2. Além disso, a formação de gás ocorre porque o peróxido de hidrogênio, H2O2, e o oxigênio do MnO4– são convertidos em H2O e O2.
Caso você precise balancear essa reação redox, também conseguimos com o ChemPy. Vamos obter a equação global balanceada das seguintes semi-reações:
Para isso passamos os reagentes e os produtos das respectivas semi-reações para a classe Equilibrium:
from chempy import Equilibriumsr_red = Equilibrium({'MnO4-': 1, 'H+': 8, 'e-': 5}, {'Mn+2': 1, 'H2O': 4})
sr_ox = Equilibrium({'H2O2': 1}, {'O2': 1, 'H+':2, 'e-':2})Agora precisamos eliminar os elétrons e multiplicar cada reação pelos seus respectivos coeficientes para obter a equação global balanceada:
coeff = Equilibrium.eliminate([sr_red, sr_ox], 'e-')
coeff[2, 5]
redox = sr_red * coeff[0] + sr_ox * coeff[1]
redoxCinética química
Além de balanceamento, o Chempy é muito útil no estudo de cinética química.
A equação de Arrhenius fornece a dependência da constante de velocidade com a temperatura como:
k = A \exp \left( -\frac{E_a}{RT} \right)Tomando o logaritmo natural da equação de Arrhenius e reorganizando a equação, temos a forma de uma equação linear:
\ln k = - \frac{E_a}{R}\frac{1}{T} + \ln AO ChemPy possibilita obter a energia de ativação e o parâmetro A utilizando a função fit_arrhenius_equation. Vamos começar importando a função:
from chempy.kinetics.arrhenius import fit_arrhenius_equationAgora vamos passar um conjunto de valores de temperatura em Kelvin, de constante de velocidade em L/(mol.s) e erros da constante de velocidade. Para isso, vamos importar a biblioteca NumPy a fim de criar os arrays e para usar posteriormente as funções logaritmo natural (log) e exponencial (exp):
import numpy as nptemperatures = np.array((1125, 1053, 1001, 838)) # K
k_values = np.array((11.59, 1.67, 0.380, 0.0011)) # L / (mol.s)
k_errors = np.array((1E-2, 1E-2, 1E-3, 1E-5))Passando as variáveis temperatures, k_values e k_errors para a função fit_arrhenius_equation vamos obter os valores da energia de ativação e do parâmetro A:
fit = fit_arrhenius_equation(temperatures, k_values, kerr=k_errors)
fit/opt/anaconda/envs/chemistry/lib/python3.9/site-packages/scipy/optimize/minpack.py:828: OptimizeWarning: Covariance of the parameters could not be estimated
(array([5.13001705e+12, 2.51435472e+05]),
array([[inf, inf],
[inf, inf]]))
Internamente, fit_arrhenius_equation utiliza o método curve_fit do SciPy. O retorno são dois arrays. O primeiro com os valores dos parâmetros; o segundo com as covariâncias quando forem possíveis de serem calculadas.
Para criar as variáveis do parâmetro A e da energia de ativação vamos acessar os elementos do primeiro array, para isso é bem simples. O primeiro array é ([5.13001705e+12, 2.51435472e+05]) cujos valores podem ser acessados como segue:
A = fit[0][0]
activation_energy = fit[0][1]
print(f'A = {A:.3e} L/(mol.s) \nEa = {activation_energy:.3e} J/mol')A = 5.130e+12 L/(mol.s) Ea = 2.514e+05 J/mol
Também podemos obter a energia de ativação e o parâmetro A com a representação gráfica de \ln k vs \frac{1}{T}.
Caso queira ver como o gráfico foi gerado com a biblioteca Matplotlib veja esse repositório.
Sistemas em equilíbrio
O ChemPy também permite efetuar cálculos de pH. Imagine um exemplo em que precisamos descobrir o pH de uma solução 0,015 mol/L de acetato.
Vamos começar criando um sistema com as equações de equilíbrio e suas constantes:
from chempy.equilibria import EqSystemequation = 'H3CCOOH = H+ + H3CCOO-; 10**-4.756'
water_autoionization = 'H2O = H+ + OH-; 10**-14/55.5'
system = EqSystem.from_string('\n'.join((equation, water_autoionization)))Para o ChemPy conseguir resolver o sistema e fornecer as concentrações de todas as espécies em solução, precisamos passar as concentrações das espécies que são conhecidas. A concentração de acetato é de 0,015 mol/L e a concentração molecular da água é de 55,5 mol/L:
from collections import defaultdict
condition = defaultdict(float, {'H2O': 55.5, 'H3CCOO-': 0.015})
concentrations, _, _ = system.root(condition)
result = dict(zip(system.substances, concentrations))
result{'H+': 3.4177702795991742e-09,
'H2O': 55.499997074115974,
'H3CCOO-': 0.014997077533741783,
'H3CCOOH': 2.922466258210992e-06,
'OH-': 2.9258840281057227e-06}
Passando a concentração de H+ para a seguinte função, retorna o pH:
def pH(hydronium_concentration):
return -np.log10(hydronium_concentration)
print(f"{pH(result['H+']):.2f}")8.47
Também podemos obter o diagrama de espécies para o ácido acético. Para isso, precisamos das frações molares do acetato e do ácido acético. Vamos criar uma função que recebe todas as espécies do sistema e retira as espécies que não serão utilizadas, retornando um dicionário das frações molares das espécies de interesse, acetato e ácido acético:
def mole_fractions(substances):
for key in ('H+', 'OH-', 'H2O'):
substances.pop(key, None)
frac = dict()
total = sum(substances.values())
for key, value in substances.items():
frac[key] = value / total
return fracAgora vamos criar um dicionário para armazenar as frações molares das espécies de interesse e criar o range de pH. O fractions foi inicialmente criado como uma lista vazia para posteriormente armazenar os valores das frações molares para cada valor de pH.
species = [item for item in system.substance_labels() if item not in ('H+', 'OH-', 'H2O')]
fractions = defaultdict(list, {k: [] for k in species})
pH_range = np.arange(0, 14.1, 0.1)
for value in pH_range:
condition = defaultdict(float, {'H2O': 55.5, 'H+': 10**-value})
concentrations, _, _ = system.root(condition)
result = dict(zip(system.substances, concentrations))
mf = mole_fractions(result)
for key, value in zip(fractions.keys(), mf.values()):
fractions[key].append(value)
/opt/anaconda/envs/chemistry/lib/python3.9/site-packages/chempy/equilibria.py:218: UserWarning: Too much of at least one component
Podemos obter o gráfico de diagrama das espécies utilizando a biblioteca Matplotlib. Para isso, vamos criar uma figura, passar os eixos e identificar o gráfico:
fig, ax = plt.subplots()
for compound in fractions:
ax.plot(pH_range, fractions[compound],
label=f'${system.substances[compound].latex_name}$')
ax.set_xlabel('pH')
ax.set_ylabel(r'$\alpha$')
ax.grid(b=True, axis='both', which='major', linestyle='--', linewidth=1.5)
ax.minorticks_on()
ax.grid(b=True, axis='both', which='minor', linestyle=':', linewidth=1.0)
ax.set_axisbelow(True)
ax.legend()
ax.set_title('Diagrama de espécies para o ácido acético', color='dimgray')
plt.show()Conclusão
Nesse artigo, vimos algumas funcionalidades das bibliotecas Molmass, Mendeleev e ChemPy e o quanto essas ferramentas são úteis na resolução de atividades da rotina de estudantes e profissionais da área química. Vimos que podemos agilizar cálculos simples, como de massa molar, até mais complexos como de concentração de espécies e de cinética.
Este artigo faz parte da tag química aqui do site, de artigos sobre a área.
Este artigo é uma colaboração minha, Helena Benevenuto, com o Ciência Programada. Me acompanhe no LinkedIn para mais conteúdos de química.
Compartilhe este artigo em suas redes e siga o projeto Ciência Programada para sempre estar atualizado. Até a próxima.




Helena muito bom o seu artigo. Eu trabalho com química computacional no programa de pós graduação na federal do ES. Você tem mais materiais de python ligado a química? Obrigado.
Muito obrigada! Em breve vou publicar mais artigos. Enquanto isso, tem a tag química do site: https://cienciaprogramada.com.br/tag/quimica/ com artigos de programação aplicada a química. Também tenho alguns projetos no GitHub: https://github.com/helenabenevenuto